关键词
最新主题
sigclust
Bioinformatics club :: Bioinformatics :: R :: R packages
第1页/共1页
160509

 sigclust
sigclust
用途
主要针对生物信息学数据(高维低样本)产生的聚类结果进行评估,一个基本假设就是数据满足单样本正态分布,计算聚类系数cluster index, 进而通过蒙特卡罗随机过程计算该系数的统计学显著性——p值。
根据p值评估聚类的效果以及聚类的结果是否是真实存在的簇,借鉴到二分k均值算法,还可以用来对多类的数据进行聚类操作
优点:计算速度快,特别适合生物信息学中的高维低样本数据
局限:只能对两分类的聚类结果进行评估,如果是多类就必须配对两两比较
函数
用于聚类效果的特征分析。使用2-means(K =2)cluster index的统计信息,探究这种分类是否真实存在。其通过仿真一元标准正态分布 来评估聚类效果,并通过数据来估计分布的参数
参数
x 一个矩阵或者data frame数据。行代表样本,列代表属性,数据需要标准化并不包含缺失值
nsim 模拟正态分布样本数(迭代循环次数),最终求得主要p值
nrep 2-means聚类的步数(默认=1,用以优化速度),如果labflag为0,则无效
labflag 如果p值用于一类或两类所指定的变量,用户定义的簇labflag=1,其他的labflag=0
label 如果labflag=0,label通过2-means簇产生,如果labflag=1,用户自行定义
icovest 协方差估计方式:1.软阈值方法,constrained MLE(默认)
2.样本协方差估计(当诊断失败建议使用)
3.使用原始背景噪声阈值估计(硬阈值方法
返回值
raw.data 原始数据矩阵
veigval 样本特征值向量
vsimeigval 在模拟中使用的特征值向量
simbackvar 来自数据的背景方差拟合
icovest 协方差估计方式
nsim 模拟正态分布的样本个数
simcindex 基于nsim模拟数据集的cluster index
pval 基于经验分布分位数模拟的p值
pvalnorm 基于正态分布分位数模拟的p值
xcindex 基于给定数据集的cluster index
效果测试
Iris鸢尾花数据
数据分布形式如下:
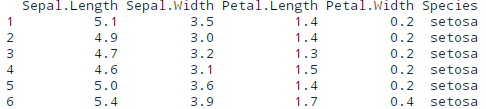
一共150个样本,分三种花型"setosa","versicolor","virginica",各50个
行代表一个样本,列代表一个属性


由此可见,1和2的分离效果明显不如1和2与2和3
主要针对生物信息学数据(高维低样本)产生的聚类结果进行评估,一个基本假设就是数据满足单样本正态分布,计算聚类系数cluster index, 进而通过蒙特卡罗随机过程计算该系数的统计学显著性——p值。
根据p值评估聚类的效果以及聚类的结果是否是真实存在的簇,借鉴到二分k均值算法,还可以用来对多类的数据进行聚类操作
优点:计算速度快,特别适合生物信息学中的高维低样本数据
局限:只能对两分类的聚类结果进行评估,如果是多类就必须配对两两比较
函数
- 代码:
sigclust(x, nsim, nrep=1, labflag=0, label=0, icovest=1)
用于聚类效果的特征分析。使用2-means(K =2)cluster index的统计信息,探究这种分类是否真实存在。其通过仿真一元标准正态分布 来评估聚类效果,并通过数据来估计分布的参数
参数
x 一个矩阵或者data frame数据。行代表样本,列代表属性,数据需要标准化并不包含缺失值
nsim 模拟正态分布样本数(迭代循环次数),最终求得主要p值
nrep 2-means聚类的步数(默认=1,用以优化速度),如果labflag为0,则无效
labflag 如果p值用于一类或两类所指定的变量,用户定义的簇labflag=1,其他的labflag=0
label 如果labflag=0,label通过2-means簇产生,如果labflag=1,用户自行定义
icovest 协方差估计方式:1.软阈值方法,constrained MLE(默认)
2.样本协方差估计(当诊断失败建议使用)
3.使用原始背景噪声阈值估计(硬阈值方法
返回值
raw.data 原始数据矩阵
veigval 样本特征值向量
vsimeigval 在模拟中使用的特征值向量
simbackvar 来自数据的背景方差拟合
icovest 协方差估计方式
nsim 模拟正态分布的样本个数
simcindex 基于nsim模拟数据集的cluster index
pval 基于经验分布分位数模拟的p值
pvalnorm 基于正态分布分位数模拟的p值
xcindex 基于给定数据集的cluster index
效果测试
Iris鸢尾花数据
数据分布形式如下:

一共150个样本,分三种花型"setosa","versicolor","virginica",各50个
行代表一个样本,列代表一个属性
- 代码:
#kmeans聚类
> clusters<-kmeans(iris[,1:4],centers=3)
> clusters[[1]]

- 代码:
#数据标准化
> normal<-function(vector){
+ (vector-mean(vector))/sd(vector)
+ }
> iris_df<-lapply(iris[,1:4],normal)
> result_matrix<-diag(rep(1,3))
> nsim <- 1000#选择迭代次数
> nrep <- 1
> icovest <- 3#选择协方差的估计方式
> for(i in 3:2){
+ for(j in 1:i-1)
+ result_matrix[i,j] <- sigclust(
+ iris[which(clusters[[1]]==i|clusters[[1]]==j),1:4],
+ nsim=nsim,
+ nrep=nrep,
+ labflag=0,
+ icovest=icovest
+ )@pvalnorm
+ }
> result_matrix

由此可见,1和2的分离效果明显不如1和2与2和3
您在这个论坛的权限:
您不能在这个论坛回复主题
» 暖贴。。。。。。。。。